研究团队利用高光谱数据和深度卷积生成对抗网络(DCGAN)来提高稻米蛋白质含量(GPC)估算的准确性。通过生成模拟数据,他们提高了模型的性能,实现了R²为 0.58 和 RRMSE 为 6.70%。该技术确定了与 GPC 相关的基因位点,包括OsmtSSB1L基因。该研究展示了将高光谱技术与 DCGAN 相结合用于高效遗传分析和优质水稻品种选择的潜力,为先进的农业实践铺平了道路。
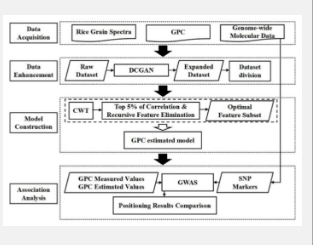
水稻 ( Oryza sativa L.) 是一种重要的农作物,为全球一半以上的人口提供粮食。对优质、富含蛋白质的水稻的需求正在上升,因此准确估算谷物蛋白质含量 (GPC) 对于培育优良品种至关重要。尽管 GWAS 等基因组工具取得了进展,但传统的表型分析仍然劳动密集且成本高昂,造成了瓶颈。光学和光谱成像的最新发展提供了高通量表型分析解决方案。然而,小型和不平衡的数据集限制了模型的性能和泛化。
2024 年 5 月 29 日在Plant Phenomics上发表的一项研究 (DOI:10.34133/plantphenomics.0200)旨在通过使用 DCGAN 生成模拟数据、提高 GPC 模型准确性并探索基因解剖潜力来解决这些问题。
该研究利用高光谱数据和 DCGAN 改进了水稻 GPC 的估算。原始和归一化光谱数据揭示了对 GPC 分析至关重要的不同吸收特征。DCGAN 在 8,000 个时期后生成的模拟数据与测量数据非常吻合,提高了模型准确性。使用这些特征的偏最小二乘回归 (PLSR) 模型实现了较高的验证准确率(R 2 = 0.58,RRMSE = 6.70%)。此外,使用模拟数据进行全基因组关联研究 (GWAS) 分析发现了重要的 SNP,包括与谷物储存蛋白相关的OsmtSSB1L基因。这种方法展示了高泛化 GPC 模型的潜力,有助于水稻品种的高级遗传分析和育种。
该研究首席研究员郑恒彪介绍说:“该研究为基于高光谱技术进行水稻表型性状的高效遗传研究提供了一种新技术。”
综上所述,本研究开发了一种使用 DCGAN 通过高光谱数据增强水稻 GPC 估计的方法。该方法展示了将 DCGAN 与高光谱技术相结合以改进作物表型和遗传分析的潜力。展望未来,在不同的生态地点和更广泛的数据集上进一步完善和验证将增强该方法的稳健性和适用性,为更精准、更有效地培育优质水稻品种铺平道路。
免责声明:本文由用户上传,如有侵权请联系删除!